 Seite drucken
Seite drucken
 Entscheidung als PDF runterladen
Entscheidung als PDF runterladen
 Seite drucken
Seite drucken
 Entscheidung als PDF runterladen
Entscheidung als PDF runterladen

Der Rechtsstreit wird bis zu einer erstinstanzlichen Entscheidung der Löschungsabteilung des Deutschen Patent- und Markenamts (DPMA) über den Löschungsantrag der Beklagten zu 1) gegen das Gebrauchsmuster DE A ausgesetzt.
Gründe:
2I.
3Die Klägerin verfolgt im Wege der Stufenklage aus einem Gebrauchsmuster gegen die Beklagten zunächst Ansprüche auf Auskunftserteilung, Rechnungslegung und sodann, nach einer nötigenfalls erforderlichen Abgabe einer eidesstattlichen Versicherung über die Richtigkeit der erteilten Informationen, auf Zahlung von der Höhe nach jeweils noch zu bestimmender Entschädigung und zu bestimmendem Schadensersatz.
4Die Klägerin ist alleinverfügungsberechtigte und eingetragene Inhaberin des deutschen Gebrauchsmusters DE A(Anlage KB2, im Folgenden: Klagegebrauchsmuster). Das Klagegebrauchsmuster wurde unter dem 11. Dezember 2015 unter Inanspruchnahme einer Priorität vom 12. Dezember 2014 (PCT/EP2014/003334) angemeldet. Die Erteilung erfolgte am 25. Januar 2022. Der Hinweis auf die Erteilung wurde am 3. März 2022 bekannt gemacht. Die Beklagte zu 1) hat am 11. November 2022 beim DPMA einen Löschungsantrag gegen das Klagegebrauchsmuster eingereicht, über den bislang keine Entscheidung ergangen ist.
5Gemäß dem Hilfsantrag der Beklagten ist der Rechtsstreit bis zu einer erstinstanzlichen Entscheidung über den Löschungsantrag auszusetzen, weil Zweifel an der Schutzfähigkeit des Klagegebrauchsmusters bestehen.
6II.
71.
8Der Klagegebrauchsmusteranspruch 1 lässt sich wie folgt gliedern:
91. Artifizielles Nukleinsäuremolekül, umfassend
101.1 mindestens einen offenen Leserahmen (ORF); und
111.2 eine 3'-untranslatierte Region (3'-UTR), umfassend
121.2.1 mindestens zwei separate Poly(A)-Sequenzen,
131.2.2 wobei eine Poly(A)-Sequenz eine Sequenz aus 20 bis 400 Adenin-Nukleotiden ist und
141.2.3 wobei die Gesamtzahl an Adenin-Nukleotiden, die von den mindestens zwei separaten Poly(A)-Sequenzen umfasst werden, mindestens 70 beträgt,
151.3 wobei der G/C-Gehalt des offenen Leserahmens gegenüber dem offenen Leserahmen des Wildtyps erhöht ist,
161.4 wobei das artifizielle Nukleinsäuremolekül ein mRNA-Molekül ist, das mindestens einen offenen Leserahmen aufweist, der für ein Antigen kodiert, das von einem mit einer Infektionskrankheit assoziierten viralen Pathogen abgeleitet ist,
171.5 zur Verwendung als Medikament, zur Verwendung als Vakzine oder zur Verwendung in der Gentherapie,
181.6 wobei das artifizielle Nukleinsäuremolekül mit einer kationischen oder polykationischen Verbindung assoziiert oder komplexiert ist, und
191.7 wobei das artifizielle Nukleinsäuremolekül, das Medikament oder die Vakzine intramuskulär verabreicht wird.
202.
21Die Parteien streiten zu Recht letztlich nur über die Schutzfähigkeit des Klagegebrauchsmusters. Die Klägerin hat substantiiert die Verletzung des Klagegebrauchsmusters vorgetragen, insbesondere auch diejenige des Merkmals 1.2 i.V.m. Merkmal 1.2.1, wonach eine 3‘-untranslatierte Region (3‘-UTR) mindestens zwei separate Poly(A)-Sequenzen umfasst. Nach derzeitiger Auffassung der Kammer sind diese Merkmale dahin zu verstehen, dass eine Poly(A)-Sequenz sowohl unmittelbar ein Bestandteil der 3’UTR, als auch an diese angefügt sein kann. In technischer Hinsicht begründet diese Anordnung keinen Unterschied. In der mündlichen Verhandlung haben sich die Beklagten nicht gegen diese zunächst als vorläufig unterbreitete Auffassung der Kammer gewendet.
223.
23Im Zentrum der Schutzfähigkeit steht zu Recht nur die Frage des erfinderischen Schritts.
24a.
25Nach § 19 S. 1 GebrMG steht es im pflichtgemäßen Ermessen des Verletzungsgerichts, ob es den Rechtsstreit aussetzt, wenn ein Löschungsverfahren anhängig ist. Das Verletzungsgericht hat den Rechtsstreit gemäß § 19 S. 2 GebrMG auszusetzen, falls es das Gebrauchsmuster für unwirksam hält.
26Die Kammer hält das Klagegebrauchsmuster vorliegend zwar nicht für unwirksam, hält allerdings eine Aussetzung aufgrund der Ausübung pflichtgemäßen Ermessens für angezeigt, § 19 S. 1 GebrMG. Bei der in diesem Rahmen anzustellenden Abwägung findet nicht der strenge Maßstab des Patentrechts Anwendung, sodass nicht der wahrscheinliche Erfolg des Löschungsantrags dargetan sein muss. Erforderlich – aber für eine Aussetzung auch hinreichend – ist es, wenn an der Schutzfähigkeit begründete Zweifel bestehen (vgl. OLG Düsseldorf, BeckRS 2014, 1152; OLG Düsseldorf, Beschluss vom 11. Juni 2018 – 15 W 30/18 –, juris; Cepl/Voß, Prozesskommentar Gewerblicher Rechtsschutz und Urheberrecht, 3. Aufl., § 148, Rn. 186). Im Gegenzug müsste für eine Verurteilung aus einem Gebrauchsmuster das Verletzungsgericht vom Rechtsbestand des Schutzrechts überzeugt sein. Dieser müsste positiv festgestellt werden können (vgl. LG Mannheim, BeckRS 2016, 16676).
27b.
28Das ist vorliegend nicht der Fall. Nach Ansicht der Kammer sprechen berechtigte Gründe gegen die Schutzfähigkeit des Klagegebrauchsmusters.
29aa.
30Gem. § 13 Abs. 1 GebrMG wird durch die Eintragung der Gebrauchsmusterschutz nicht begründet, soweit gegen den als Inhaber Eingetragenen für jedermann ein Anspruch auf Löschung, § 15 Abs. 1, Abs. 3 GebrMG, besteht. Dies ist insbesondere dann der Fall, wenn der Gegenstand des Gebrauchsmusters nach den §§ 1 bis 3 nicht schutzfähig ist. Gem. § 1 Abs. 1 GebrMG sind solche Erfindungen einem Gebrauchsmusterschutz zugänglich, die neu sind und auf einem erfinderischen Schritt beruhen.
31Nach der Rechtsprechung des BGH handelt es sich bei der Beurteilung der erfinderischen Tätigkeit um eine Rechtsfrage, die mittels wertender Würdigung der tatsächlichen Umstände zu beurteilen ist, die unmittelbar oder mittelbar geeignet sind, etwas über die Voraussetzungen für das Auffinden der erfindungsgemäßen Lösung auszusagen. Dies gilt gleichermaßen für die Beurteilung des erfinderischen Schritts im Gebrauchsmusterrecht. Wie im Patentrecht ist maßgeblich, ob der Stand der Technik am Prioritätstag dem Fachmann den Gegenstand der Erfindung nahegelegt hat. Dies erfordert zum einen, dass der Fachmann mit seinen durch seine Ausbildung und berufliche Erfahrung erworbenen Kenntnissen und Fähigkeiten in der Lage gewesen ist, die erfindungsgemäße Lösung des technischen Problems aus dem Vorhandenen zu entwickeln. Dies allein genügt jedoch nicht, um den Gegenstand der Erfindung als nahegelegt anzusehen. Hinzukommen muss vielmehr zum anderen, dass der Fachmann Grund hatte, den Weg der Erfindung zu beschreiten. Dazu bedarf es in der Regel über die Erkennbarkeit des technischen Problems hinausreichender Anstöße, Anregungen, Hinweise oder sonstiger Anlässe. Denn nur dann kann die notwendigerweise ex post getroffene richterliche Einschätzung, dass der Fachmann ohne erfinderisches Bemühen zum Gegenstand der Erfindung gelangt wäre, in einer Weise objektiviert werden, die Rechtssicherheit für den Schutzrechtsinhaber wie für seine Wettbewerber gewährleistet. Dabei lässt sich keine allgemeine, vom jeweiligen Streitfall losgelöste Aussage darüber treffen, in welchem Umfang und mit welcher Konkretisierung der Fachmann Anregungen im Stand der Technik benötigt, um eine bekannte Lösung in bestimmter Weise weiterzuentwickeln. Es handelt sich vielmehr um eine Frage des Einzelfalls, deren Beantwortung eine Gesamtbetrachtung aller maßgeblichen Sachverhaltselemente erfordert. Dabei sind nicht etwa nur ausdrückliche Hinweise an den Fachmann beachtlich. Vielmehr können Eigenarten des in Rede stehenden technischen Fachgebiets, insbesondere Ausbildungsgang und Ausbildungsstand der auf diesem Gebiet tätigen Fachleute zum Prioritätszeitpunkt und die auf dem technischen Fachgebiet übliche Vorgehensweise von Fachleuten bei der Entwicklung von Neuerungen ebenso eine Rolle spielen wie technische Bedürfnisse, die sich aus der Konstruktion oder der Anwendung des in Rede stehenden Gegenstands ergeben, nicht-technische Vorgaben, die geeignet sind, die Überlegungen des Fachmanns in eine bestimmte Richtung zu lenken, und umgekehrt Gesichtspunkte, die dem Fachmann Veranlassung geben könnten, die technische Entwicklung in eine andere, von der Erfindung wegweisende Richtung voranzutreiben (BGH, GRUR 2012, 378 – Installiereinrichtung II).
32Bei der Prüfung auf erfinderische Tätigkeit sind auch Handlungsweisen zu berücksichtigen, die dem Fachmann deshalb nahegelegt waren, weil sie am Prioritätstag zum Standard-Repertoire gehörten. Insbesondere bei Maßnahmen, die nicht dazu dienen, eine für den in Rede stehenden Stoff spezifische Wirkung hervorzurufen, sondern dazu, unerwünschte Wirkungen allgemeiner Art zu verhindern oder die von dem Stoff ausgehenden Wirkungen in örtlicher oder zeitlicher Hinsicht einzugrenzen, wird es häufig vom Zufall abhängen, ob die Anwendung dieser Maßnahme auch und gerade für einen bestimmten Stoff schriftlich belegt werden kann. Ein solcher Beleg ist jedenfalls dann nicht erforderlich, wenn feststeht, dass der Fachmann die in Rede stehende Maßnahme am Prioritätstag als generelles Mittel für eine Vielzahl von Anwendungsfällen in Betracht gezogen hat und dass keine besonderen Umstände vorlagen, die eine Anwendung in der konkret zu beurteilenden Konstellation als nicht möglich oder untunlich erscheinen ließen (GRUR 2014, 461– Kollagenese I).
33bb.
34Ausgehend von diesen Grundsätzen hat die Kammer Zweifel, dass die im Klagegebrauchsmuster zum Ausdruck kommende technische Lehre auf einem erfinderischen Schritt beruht.
35Zwischen den Parteien wird diskutiert, ob ausgehend von der Druckschrift WO B(Anlage B06-Ast 17; im Folgenden: WO‘B) gegebenenfalls in Kombination mit der Veröffentlichung Milone et al. (Anlage B2; B06-Ast 07) Anlass für den Fachmann bestand, im Stand der Technik vorbekannte mRNA-Strukturen in Richtung auf eine anspruchsgemäße Variante zu entwickeln, und ob eine solche Ausgestaltung nahelag.
36(1)
37Die WO‘B bildet den nächstliegenden Stand der Technik und offenbart eine Nukleinsäuresequenz, umfassend oder kodierend für eine kodierende Region, die für mindestens ein Peptid oder Protein kodiert, umfassend ein pathogenes Antigen oder ein Fragment, eine Variante oder ein Derivat davon, mindestens einen Histon-Stem-Loop und eine Poly(A)-Sequenz oder ein Polyadenylierungssignal. Zudem wird die Verwendung der Nukleinsäure zur Erhöhung der Expression des genannten kodierten Peptids oder Proteins offenbart sowie dessen Verwendung zur Herstellung einer pharmazeutischen Zusammensetzung, insbesondere eines Impfstoffes, z. B. zum Einsatz in der Behandlung von Infektionskrankheiten. Die Druckschrift beschreibt außerdem ein Verfahren zur Erhöhung der Expression eines Peptids oder Proteins, umfassend ein pathogenes Antigen oder ein Fragment, eine Variante oder ein Derivat davon, unter Verwendung der Nukleinsäure umfassend oder kodierend für einen Histon-Stem-Loop und eine Poly(A)-Sequenz oder ein Polyadenylierungssignal (vgl. Anlage Ast 17, S. 1).
38Die WO‘B sieht ihre Aufgabe in der Bereitstellung zusätzlicher und/oder alternativer Verfahren zur Erhöhung der Expression eines kodierten Proteins, vorzugsweise über eine weitere Stabilisierung der mRNA und/oder eine Erhöhung der translationalen Effizienz einer solchen mRNA in Bezug auf die vorbekannten Nukleinsäuren zur Verwendung in einer genetischen Impfung in der therapeutischen oder prophylaktischen Behandlung von Infektionskrankheiten (Anlage Ast 17, S. 8).
39Hierzu wird eine Nukleinsäuresequenz vorgeschlagen, umfassend oder kodierend für a) eine kodierende Region, die für mindestens ein Peptid oder Protein kodiert, umfassend ein pathogenes Antigen oder ein Fragment, eine Variante oder ein Derivat davon; b) mindestens einen Histon-Stem-Loop, und c) eine Poly(A)-Sequenz oder ein Polyadenylierungssignal, bevorzugt zur Erhöhung der Expression des genannten Peptids oder Proteins (Anlage Ast 17, S. 8).
40Im Hinblick auf die Lehre des Klagegebrauchsmusters sind jedenfalls die Merkmalsgruppe 1.2.1-1.2.3 sowie Merkmal 1.6 nicht durch die WO‘B offenbart. Der Fachmann hatte aber Anlass, von der Lehre der WO‘B weiterzugehen und sie um die fehlenden Merkmale zu ergänzen.
41Denn es spricht viel dafür, dass der Fachmann aus der WO‘B die Sequenz „H1N1(PR8)-AG-A64-C30-histoneSL“ als Ausgangspunkt seiner Überlegungen wählen würde, da er erkennt, dass mit Blick auf dieses mRNA-Konstrukt Spielraum für Verbesserungen besteht.
42In der Figur 19 ist diese Sequenz dargestellt:
43![]()
Unterschiedliche Konstrukte von mRNA und deren Wirkung werden in der WO‘B in der Figur 21 gezeigt. Es heißt dazu auf Seite 104:
45„Figur 21 zeigt, dass die Kombination von Poly(A) und HistonSL die Protein-Expression von mRNA unabhängig von ihrer Reihenfolge erhöht. Es wurde der Effekt der Poly(A)-Sequenz, HistonSL, der Kombination aus Poly(A) und HistonSL und ihre Reihenfolge auf die Luciferase-Expression von mRNA untersucht. Daher wurden unterschiedliche mRNAs in HeLa-Zellen lipofektiert. Es wurden die Luciferase-Spiegel nach 6, 24 und 48 Stunden nach dem Beginn der Transfektion gemessen. Sowohl eine A64 Poly(A)-Sequenz, also auch die HistonSL führen zu vergleichbaren Luciferase-Spiegeln. Eine Erhöhung der Länge der Poly(A)-Sequenz von A64 auf A120 oder auf A300 erhöht den Luciferase-Spiegel moderat. Im Gegensatz dazu erhöht die Kombination aus Poly(A) und HistonSL den Luciferase-Spiegel sehr viel weiter als eine Verlängerung der Poly(A)-Sequenz. Die Kombination von Poly(A) und HistonSL agiert synergistisch, da sie den Luciferase-Spiegel um ein Vielfaches über dem Niveau erhöht, das mit je einem der einzelnen Elemente beobachtet wurde. Es wird ein synergistischer Effekt der Kombination aus Poly(A) und HistonSL unabhängig von der Reihenfolge von Poly(A) und HistonSL beobachtet und unabhängig von der Länge der Poly(A) mit A64-HistonSL oder HistonSL-A250 mRNA. Die Daten werden graphisch als mittlere RLU ± SD für dreifache Transfektionen dargestellt. Spezifische RLU sind in Beispiel 11.3 zusammengefasst.“
46Die Figur 21 ist im Folgenden eingeblendet:
47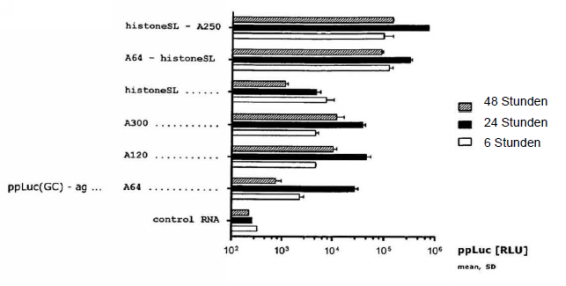
Dieser kann der Fachmann entnehmen, dass gerade die Zusammensetzungen, welche eine histonSL aufweisen, gesteigerte Wirksamkeit zeigen.
49Dabei ist - entgegen der Auffassung der Klägerin - die mRNA mit A64 und histoneSL ein passender Ausgangspunkt in den Überlegungen des Fachmanns. Die Klägerin begründet ihre Ansicht damit, dass der Fachmann nicht die kürzeste Poly(A)-Sequenz heranziehen würde, wenn er doch gerade diese verlängern bzw. eine zweite Poly(A)-Sequenz vorsehen wolle. Allerdings ist kein Grund ersichtlich, dass der Fachmann als Ausgangspunkt eine hinsichtlich der Proteinexpression schlechtere Sequenz auswählen würde, obwohl er Kenntnis von Strukturen mit einer besseren Wirkung hat. HistoneSL-A250 wäre daher nicht als besserer Ausgangspunkt in Betracht gekommen. Denn aus der Verlängerung der mRNA-Strukturen nur mit Adenin-Nukleotiden erkennt der Fachmann, dass A300 gegenüber A120 keine bessere Proteinexpression mehr zur Verfügung stellen konnte. Die dabei eintretende Expressionssteigerung fällt geringer aus als diejenige von A64 mit histoneSL gegenüber Sequenzen mit lediglich Adenin-Nukleotiden. Der Fachmann hatte deshalb Grund zu der Annahme, dass auch bei histoneSL-A250 keine Steigerungschancen mehr verbleiben.
50Ebenso wenig ist zu erkennen, dass der Fachmann eher an Merkmalen wie den UTR-Elementen, der 5‘-Cap Struktur oder Formulierung etc. Verbesserungen vornehmen würde und nicht aber an denjenigen Elementen, die den synergistischen Effekt hervorbringen. Technische Argumente führt die Klägerin hierzu keine an. Insbesondere ist nicht bekannt, inwieweit diese vor dem 3‘-Ende gelegenen Bestandteile der mRNA einen Beitrag zur Wirkung leisten und sich positiv auf die Stabilität sowie Proteinexpression auswirken
51Vielmehr hat der Fachmann aufgrund der in der Figur 21 dargestellten positiven Ergebnisse einen Anhalt, an der Sequenz „H1N1(PR8)-AG-A64-C30-histoneSL“ Veränderungen vorzunehmen. Er wäre bestrebt, ausgehend davon weitere Verbesserungen anzustreben. Der Fachmann dürfte gerade anhand der in der Figur 21 gezeigten Ergebnisse sehen, dass sich das Vorhandensein von histoneSL vorteilhaft auswirkt, weshalb er dieses daher auch anderen Nukleinsäuresequenzen zugrunde legen würde. Dies gilt umso mehr, als der Fachmann aus den Untersuchungen den Hinweis erhalten hat, dass das Anfügen eines Elements wie z.B. histoneSL die Eigenschaften der mRNA positiv verändert. Der Fachmann würde nicht versuchen, Bestandteile innerhalb der mRNA zu verlängern.
52Hiervon ausgehend war es für den Fachmann naheliegend, weitere Adenin-Nukleotide anzufügen und zwar in einer separaten Poly(A)-Sequenz. Ferner kannte der Fachmann die intramuskuläre Verabreichungsform für mRNA.
53(2)
54Schon die WO‘B gibt dem Fachmann Anreize, die darin erörterte mRNA-Sequenz weiter zu verändern. Der Fachmann erkennt, dass die von der WO‘B offenbarten Verbindungen keine abschließende Lösung darstellen, die WO‘B vielmehr einen Anlass bietet, weitere Veränderungen vorzunehmen.
55Auf Seite 61 der WO‘B wird die Möglichkeit, einzelne Elemente einer mRNA-Struktur innerhalb derselben zu wiederholen, ausdrücklich angesprochen. Dort heißt es:
56„Die Elemente b) bis c) der erfindungsgemäßen Nukleinsäure können in jeglicher Reihenfolge in der erfindungsgemäßen Nukleinsäuresequenz vorkommen, d.h. die Elemente a), b) und c) können in der Reihenfolge a), b) und c) oder a), c) und b) von der 5' bis 3'-Richtung, wobei weitere Elemente wie vorliegend beschreiben auch enthalten sein können, wie etwa eine 5'-CAP-Struktur, eine Poly(C)-Sequenz, Stabilisierungssequenzen, IHRES-Sequenzen, etc. Jedes der Elemente a) bis c) der erfindungsgemäßen Nukleinsäure, insbesondere a) in di- oder multizistronischen Konstrukten und jedes der Elemente b) und c), bevorzugter Element b) kann auch mindestens einmal in der erfindungsgemäßen Nukleinsäure wiederholt werden, bevorzugt zwei Mal oder öfter. Beispielhaft kann die erfindungsgemäße Nukleinsäure ihre Sequenzelemente a), b) und optional c) z. B. in der folgenden Reihenfolge zeigen: 5' – kodierende Region – Histon-Stem-Loop – Poly(A)-Sequenz - 3'; oder 5' – kodierende Region – Histon-Stem-Loop – Polyadenylierungssignal - 3'; oder 5' – kodierende Region – Poly(A)-Sequenz – Histon-Stem-Loop - 3'; oder 5' – kodierende Region – Polyadenylierungssignal – Histon-Stem-Loop - 3'; oder 5' – kodierende Region – kodierende Region – Histon-Stem-Loop – Polyadenylierungssignal - 3'; oder 5' – kodierende Region – Histon-Stem-Loop – Histon-Stem-Loop – Poly(A)-Sequenz - 3'; oder 5' – kodierende Region – Histon-Stem-Loop – Histon-Stem-Loop – Polyadenylierungssignal - 3'; etc.“
57Hier wird deutlich gemacht, dass die Sequenzelemente a) bis c) wiederholt werden können. In den darauffolgenden Beispielen ist zwar keine Variante enthalten, die neben einem doppelten Histon-Stem-Loop auch eine zweite Poly(A)-Sequenz vorsieht. Hieraus kann allerdings nicht geschlossen werden, dass die genannten Varianten abschließend gemeint sind. Die durch die Bezeichnung „etc.“ am Ende wird verdeutlicht, dass die Offenbarung auf die genannten Varianten nicht beschränkt ist. Der Fachmann entnimmt ferner der Figur 21, dass sich genau die Kombination von Poly(A)-Sequenz und Histon-Stem-Loop in der Proteinexpression vorteilhaft ausgewirkt hat. Er hat daher Grund für die Annahme, dass sich eine Verdopplung dieser beiden Elemente weiterhin vorteilhaft auswirken kann.
58Überdies erlangt der Fachmann auch durch weitere, von den Beklagten im Laufe des Verletzungsverfahrens angeführte Dokumente Hinweise auf stabilere Poly(A)-Sequenzen sowie auf unterbrochene Poly(A)-Strukturen.
59So offenbart die US C(Anlage B6-Ast 18; im Folgenden: US‘C) die Unterbrechung einer Poly(A)-Sequenz durch andere Nukleotide als Adenine. Die US‘C bezieht sich auf Verfahren zur Veränderung eukaryotischer, vorzugsweise Säugetier-mRNA, die zu deren Stabilisierung gegen Nukleasen führen und deren Verwendung in der Sense-RNA-Therapie zur Bereitstellung eines interessierenden Proteins ermöglichen. Zur Erreichung der Stabilisierung schlägt die US‘C verschiedene Weisen vor (vgl. Abs. [0053]). Neben dem Basenabbau, der chemischen Modifikation kommt insbesondere auch die Verlängerung des Poly(A)-Schwanzes in Betracht (ab Abs. [0099] ff.). Dazu ist ausgeführt:
60„[0099] In anderen Ausführungsformen der Erfindung kann die Stabilisierung der mRNA durch eine Erhöhung der Länge des Poly-A-Schwanzes erreicht werden. Es wird angenommen, dass der Poly-A-Schwanz natürliche Boten und synthetische Sense-RNAs stabilisiert.
61[0100] In einer Ausführungsform kann einem mRNA-Molekül, z.B. einem synthetischen RNA-Molekül, ein langer Poly-A-Schwanz hinzugefügt werden, wodurch die RNA noch stabiler wird. Poly-A-Schwänze können mit einer Reihe von im Stand der Technik anerkannten Verfahren hinzugefügt werden. Beispielsweise können lange Poly-A-Schwänze mithilfe der Poly-A-Polymerase an synthetische oder in vitro transkribierte RNA angehängt werden (Yokoe und Meyer. Nature Biotechnology 14, 1252-1256 (1996)). Lange Poly-A-Schwänze können auch durch einen Transkriptionsvektor kodiert werden, allerdings können lange Wiederholungen während der bakteriellen Vermehrung instabil sein. Die Verwendung von Bakterienstämmen mit Mutationen in Rekombinationsenzymen (wie sie z. B. von Stratagene im Handel erhältlich sind) kann dieses potenzielle Problem verringern. Darüber hinaus können Poly-A-Schwänze durch eine direkt von PCR-Produkten erfolgende Transkription hinzugefügt werden, wie in den angehängten Beispielen beschrieben. Poly-A kann ferner mittels RNA-Ligase an das 3‘- Ende einer Sense-RNA ligiert werden (siehe z. B. Molecular Cloning A Laboratory Manual, 2. Aufl., hrsg. von Sambrook, Fritsch und Maniatis (Cold Spring Harbor Laboratory Press: Ausgabe 1989 oder 1991)). Modifizierte Poly-A-Schwänze können auch mit einem Splint-Ligationsverfahren hinzugefügt werden (Biochemie. 1994. 76:1235).
62[0101] Wie bereits erwähnt, können Phosphorothioat-Bindungen bzw. andere stabilisierende Modifikationen der RNA zur weiteren Stabilisierung eines mRNA-Moleküls in einen Poly-A-Schwanz eingebaut werden. Solche modifizierten Poly-A-Schwänze können mittels Poly-A-Polymerase an Sense-mRNA-Moleküle angehängt werden. In einer weiteren Ausführungsform können andere, stärker modifizierte Analoga von A oder anderen Basen in einen Poly-A-Schwanz eingebaut werden (z. B. 2‘-O-Methyl- oder -Methylphosphonat-modifizierte). In einer Ausführungsform der Erfindung können stromabwärts des Poly-A-Schwanzes weitere Modifikationen vorgenommen werden, um die Poly-A-Bindungsstellen zu erhalten und 3‘-Exonukleasen weiter zu blockieren.
63[0102] In einer Ausführungsform beträgt die Länge des Poly-A-Schwanzes mindestens etwa 90 Nukleotide, in einer anderen Ausführungsform beträgt die Länge des Poly-A-Schwanzes mindestens etwa 200 Nukleotide.“
64Da die eingefügten Nukleotide nicht der eigentlichen Poly(A)-Sequenz zugerechnet werden, liegt eine Modifikation, mithin eine Unterbrechung, einer Poly(A)-Sequenz vor. Die gegen das Verständnis des zitierten Abs. [0101] geäußerte Kritik der Klägerin, dass es sich bei dem Zusatz „oder andere Basen“ um einen Schreibfehler handele, welchen der Fachmann unberücksichtigt lasse, ist nach Einschätzung der Kammer zumindest nicht hinreichend, um die Argumente für die Heranziehung dieser Druckschrift zu zerstreuen. Denn eine nähere Begründung für ihre Auffassung hat die Klägerin in technischer Hinsicht nicht angeführt. Damit sind der US‘C Erkenntnisse über mRNA-Konstrukte zu entnehmen, die mehr als A64 enthalten, welche außerdem nicht in einem Strang vorliegen. Dadurch wird der Fachmann in den Bestrebungen unterstützt, die Ausgangssequenz von A64 mit HistoneSL um Adenin-Nukleotide zu verlängern, ohne das Erfordernis, diese unmittelbar an der A64 anschließen zu müssen.
65Ähnliches ergibt sich aus der Veröffentlichung Khanam et al, „Poly(A)-Binding Protein Binds to A-Rich Sequences via RNA-Binding Domains 1+2 and 3+4“, RNA Biology 3:4, 170-177, October/November/December 2006 (Anlage B3).
66Khanam et al. beschäftigt sich mit der Frage, wie das Poly(A)-Bindungsprotein (PABP) an nicht-Protein-kodierende mRNA-Sequenzen bindet. In Untersuchungen kam heraus, dass kürzere RNA kein ideales Substrat für Bindungsstudien ist, um die Interaktion mit mRNAs zu modellieren. Die Bindungsaffinität des PABP war bei adenosinreichen mRNAs erhöht.
67Auf Seite 170 heißt es:
68„Es ist bekannt, dass PABP mit mRNAs durch Bindung an ihren Poly(A)-Schwanz interagiert, wodurch die mRNA-Translation stabilisiert und reguliert wird.“
69Auf Seite 174 wird ausgeführt:
70„Zur weiteren Untersuchung der Bedeutung der Oligo(A)-Länge in Bindungsstudien wurden ähnliche Studien mit Oligo(A)65 durchgeführt. Die Bindungsaffinität von Oligo(A)65 entsprach der von BC1- und BC200-RNA und Mini-mRNA/A98. Die Reihenfolge der Bindungsaffinitäten von Oligo(A)65 an Protein war die Folgende: RRMs 1+2 > PABP voller Länge > RRMs 3+4 > RRMs 2+3. Während die RRMs 3+4 Oligo(A)25 nicht effizient binden, banden sie an Oligo(A)65 mit sehr hoher Affinität (Fig. 3B), vergleichbar mit der von BC1-RNA oder BC200-RNA. Daraus wird geschlossen, dass die Oligo(A)-Länge eine wichtige Rolle bei der Interaktion mit PABP spielt, was auf die Bedeutung der Poly(A)-Länge in mRNAs oder anderen RNAs übertragen werden könnte, unabhängig davon, ob das Oligo(A) durch andere Nukleotide unterbrochen wird.“
71Die Ausführungen enthalten Aussagen zur Stabilität einer mRNA, die in Zusammenhang mit der Anzahl an Adenin-Nukleotiden steht. Es kann dahinstehen, ob das eigentliche Untersuchungsziel von Khanam et al. auf etwas anderes als die Stabilität von mRNA gerichtet ist, und zwar das Bindungsverhalten von dem Poly(A)-Bindeprotein PABP, solange jedenfalls dabei auch Feststellungen über die Vorteilhaftigkeit von längeren Poly(A)-Sequenzen getroffen werden konnten. Genau das ist der Fall, wie die zitierte Passage zeigt. Außerdem kann der Fachmann diesem Absatz entnehmen, dass die Oligo(A)-Sequenz durch andere Nukleotide unterbrochen war. Demnach waren im Stand der Technik mRNA-Strukturen bekannt, die nicht durchgängige Poly(A)-Sequenzen enthielten.
72Der Bedeutungsgehalt von Khanam et al. ist entgegen der Auffassung der Klägerin nicht dadurch geschmälert, dass nur in-vitro Experimente im Hinblick auf das Bindungsverhalten gezeigt wurden. Selbst wenn die Durchführung der Untersuchungen in-vitro anderen Bedingungen unterliegt als eine in-vivo Verabreichung, sind diese Tests dennoch geeignet, die grundsätzliche Wirkweise aufzuzeigen. Die Klägerin hat hierzu nur pauschale andere „Faktoren“ angeführt, die sich neben den getesteten zwei Umständen auf die Protein-Expression in vivo auswirken sollen, was nicht ausreichend ist, jeglichen Rückschluss von in-vitro Experimenten auf die realen Umstände zu verbieten.
73Ausgehend von der WO‘B war der Fachmann ferner veranlasst, eine intramuskuläre Verabreichung vorzusehen. Schon aus dem Dokument WO‘B erhält er den Anhaltspunkt dafür, indem neben anderen Verabreichungsformen auch die intramuskuläre Variante angeführt wird. Unerheblich hierbei ist, dass hinsichtlich einer in vivo Verabreichung lediglich eine interdermale Abgabe erläutert wird. Denn der Fachmann verfügt über hinreichende Kenntnisse darüber, dass mRNA tatsächlich intramuskulär an einen Menschen abgegeben werden kann.
74Die Klägerin hat demgegenüber nicht aufgezeigt, dass die intramuskuläre Verabreichung strengeren/anderen Anforderungen unterläge als die interdermale Abgabe. Insoweit wäre auch allenfalls zu erwarten, dass ein Wechsel von intramuskulär auf interdermal Schwierigkeiten hinsichtlich der Verträglichkeit oÄ bereiten könnte, indes nicht im umgekehrten Fall. Es wäre an der Klägerin gewesen, konkrete Anhaltspunkte aufzuzeigen, die den Fachmann von einer intramuskulären Verabreichung abgehalten hätten. Ohne diese Hinweise ist aber davon auszugehen, dass der Fachmann auf seine Vorkenntnisse zurückgreifen wird und denjenigen Verabreichungsweg wählen wird, der für den Impfstoff geläufig ist.
75(3)
76Anregungen aus dem Stand der Technik zur Anfügung einer separaten Poly(A)-Sequenz konnte der Fachmann ferner aus Milone et al., „Characterization of deadenylation in trypanosome extracts and its inhibition by poly(A)-binding protein Pab1p“, RNA (2004), 10:448–457 (Anlage B2; B06-Ast 07) erhalten.
77Milone et al. befasst sich mit der Charakterisierung der Deadenylierung in Trypanosomextrakten und deren Inhibierung durch das Poly(A)-bindende Protein Pab1p. Die in dieser Veröffentlichung an Trypanosomen vorgenommenen Untersuchungen beruhen auf der grundlegenden Erkenntnis, dass die Steuerung der Genexpression ein grundlegendes Merkmal aller Organismen ist. Die meisten Eukaryoten regulieren die Genexpression auf verschiedenen Ebenen, von der Initiierung der Transkription bis zur Translation (vgl. S. 448). Untersuchungen haben gezeigt, dass die mRNA-Umsetzung einer der wichtigsten Steueransatzpunkte ist. Diese einleitenden Ausführungen in Milone et al. zeigen, dass lediglich für eine spezielle Art von Organismus die Funktionsweise von mRNA getestet werden soll. Trypanosomen sind parasitäre Protozoen, die viele Abweichungen vom normalen Paradigma der Steuerung der Genexpression in Eukaryoten aufweisen. Inwieweit aus diesen Untersuchungen gewonnene Erkenntnisse unverändert auf Säugetiere übertragbar sind, ist fraglich. Ein Fachmann, der sich mit dem System eines Säugetiers und darauf abgestimmte pharmazeutischen Zusammensetzungen auseinandersetzt, nimmt möglicherweise nicht ohne Weiteres Informationen über andere Organismen in den Blick.
78Hingegen finden sich in Milone et al. aber auch Passagen, die auf Übereinstimmungen in den verschiedenen Systemen von Säugetieren/Eukaryoten und Trypanosomen hindeuten, was wiederum für die Berücksichtigung dieser Veröffentlichung spricht, weil die Untersuchungen an Trypanosomen auf mRNA-Strukturen für einen Säugetier-Organismus übertragbar sein könnten. Neben dem schon eingangs erwähnten Rückgriff auf Erkenntnisse über den Säugetier-Organismus, heißt es an anderer Stelle wie folgt:
79S. 451: „Die Trypanosomen Deadenylase-Aktivitäten weisen ähnliche Merkmale wie andere bekannte Deadenylase-Aktivitäten auf.“
80S. 453: „Wir haben die Entwicklung eines in vitro mRNA-Umsetzungssystems für Trypanosomen beschrieben, das für viele der enzymatischen Schritte in den wichtigsten Umsetzungspfaden von Säugetieren und Hefe funktional ist. Diese trypanosomale Aktivität ist für Adenylatreste spezifisch und weist viele der Merkmale anderer charakterisierter Deadenylasen auf, […].“
81„In dieser Hinsicht hat die Entdeckung der trypanosomalen Deadenylierungsaktivität einen Vergleich mit bestehenden Deadenylase-Aktivitäten in anderen Eukaryoten ermöglicht. Die trypanosomale Aktivität ähnelt in ihrer Empfindlichkeit gegenüber Neomycin-B-Sulfat der PARN bei Säugetieren, unterscheidet sich aber im Erfordernis der Cap-Abhängigkeit der Aktivität.“
82Da jedenfalls aufgrund vorstehender Aussagen die Hinzuziehung der Veröffentlichung Milone et al. nicht mit Sicherheit ausgeschlossen werden und der Fachmann hieraus Anhaltspunkte auf die Möglichkeit von unterbrochenen Poly(A)-Sequenzen erhalten kann, verbleiben Zweifel an der Schutzfähigkeit des Klagegebrauchsmusters.
83(4)
84Aus den Erfahrungen des Standes der Technik lag es daher nahe, eine stabile mRNA zu entwickeln. Der Fachmann hatte daher hinreichenden Grund, mehrere Adenin-Nukleotide in getrennten Sequenzen und nicht lediglich eine lange Poly(A)-Sequenz vorzusehen und dies vor allem deshalb, weil die WO‘B schon eine Struktur aus A250 und histoneSL vorsah und deshalb die Frage nahelag, wie sich die Proteinexpression bei anderen angehängten Adenin-Nukleotiden entwickelt.
85Klepsch Wimmers Dr. Janich
86Vorsitzende Richterin Richterin am Landgericht Richter
87am Landgericht